特定代谢物检测
检测内容:
定制代谢组及靶向代谢组学,是对特定的代谢物群进行检测与分析。分析时加入了标准品的定量分析,用以对特定代谢物绝对浓度的定量检测。常规分析法有气质联用(GC-MS)和液质联用(LC-MS),我们优先利用LC-MS的方法进行代谢组分析[1],然后通过与相同分析条件下标准品的分析结果进行比对,最终完成目标代谢群定性和定量的分析。
针对不同靶向检测物,我们制备了多种标准品,可对胆汁酸、短链脂肪酸、TMAO及相关代谢物、氨基酸、脂肪酸、有机酸、黄酮类、植物激素、神经递质等进行靶向检测。
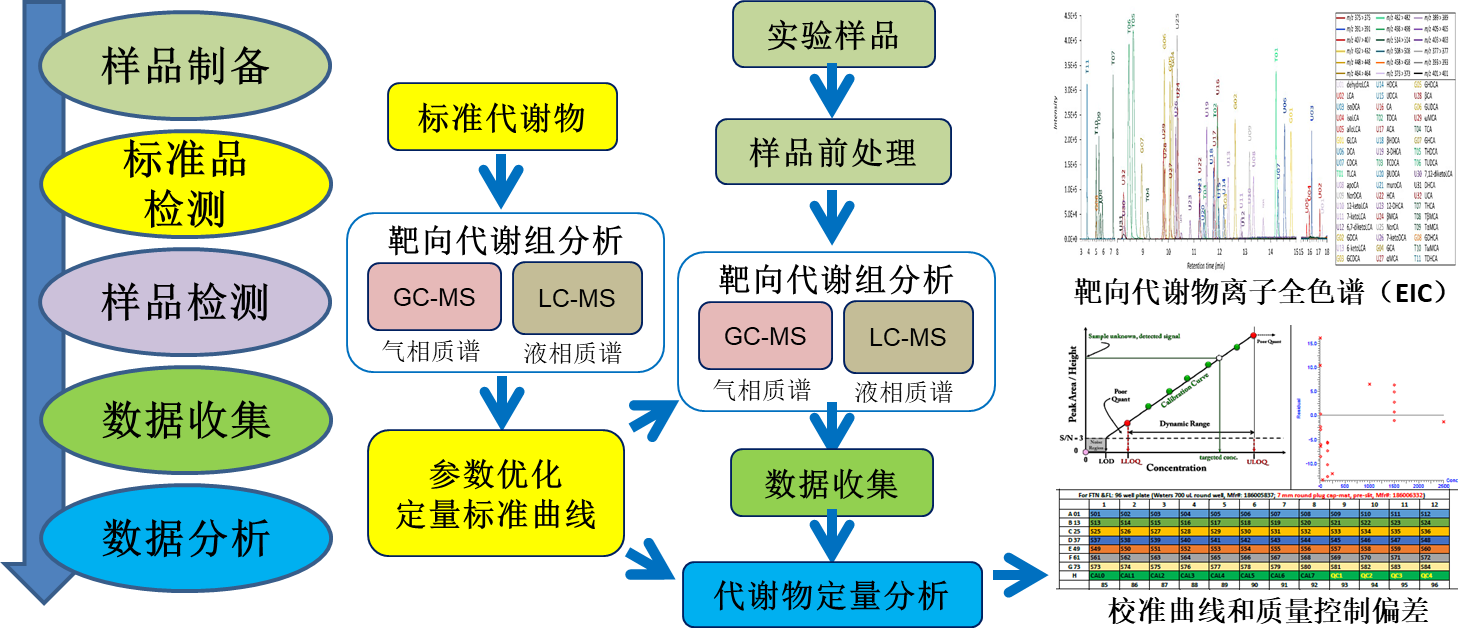
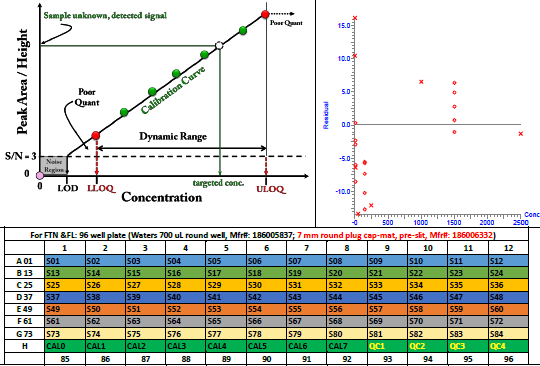
图1 校准曲线和质量控制偏差
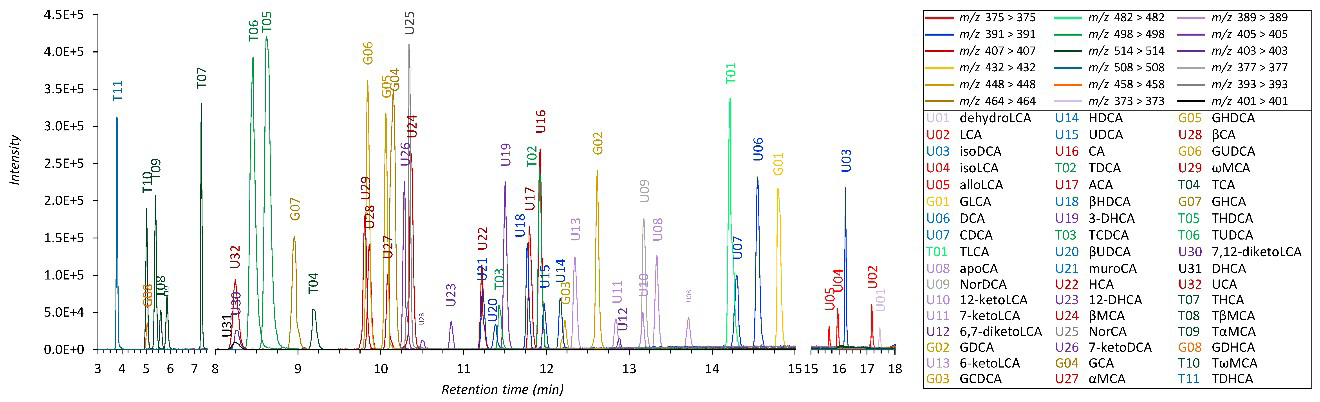
图2 所有单个胆汁酸的提取离子色谱(EIC、全谱)
表1 胆汁酸检测结果
| 序号 | 样本类型 | 样本量 | 检测平台 |
|---|---|---|---|
| 1 | 粪便 | 500mg鲜重/50mg冻干粉 | NMR |
| 1 | 粪便 | 200mg鲜重/20mg冻干粉 | GC-MS/LC-MS |
| 2 | 血清 | 500μl | NMR |
| 2 | 血清 | 200μl | GC-MS/LC-MS |
| 3 | 血浆 | 500μl | NMR |
| 3 | 血浆 | 200μl | GC-MS/LC-MS |
| 4 | 尿液 | 500μl | NMR |
| 4 | 尿液 | 200μl | GC-MS/LC-MS |
| 5 | 组织 | 500mg鲜重/50mg冻干粉 | NMR |
| 5 | 组织 | 200mg鲜重/20mg冻干粉 | GC-MS/LC-MS |
| 6 | 细胞 | 500mg鲜重/50mg冻干粉 | NMR |
| 6 | 细胞 | 200mg鲜重/20mg冻干粉 | GC-MS/LC-MS |
| 7 | 细菌 | 500mg鲜重/50mg冻干粉 | NMR |
| 7 | 细菌 | 200mg鲜重/20mg冻干粉 | GC-MS/LC-MS |
采样送样建议:
1),采样过程中注意消毒,防止微生物污染,建立好同等条件下的负对照样本。
2),取得样本后迅速放入液氮速冻;
2),无液氮情况下,迅速转入-80℃冰箱保存,期间不得反复冻融;
3),全程干冰运送。
生物学重复:
在现下的临床医学中,个体差异的影响越发明显,建议样品数量在15-30以上,培养的细胞和动物模型等样本,生物学重复数建议在6个样本以上。

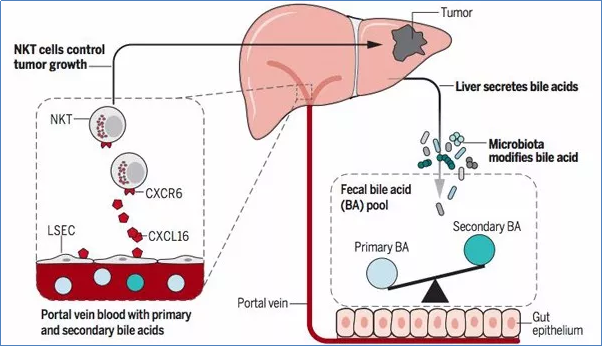
肠道微生物调控肝脏肿瘤生长思路示意图
2018 年5月,美国NIH癌症研究中心研究人员发现肠道微生物可以通过代谢产物胆汁酸特异性调节肝脏中一种免疫细胞——自然杀伤T(NKT)细胞的数量,从而调控肝脏肿瘤的生长,并将成果发表在了《Science》上。
起初,他们发现了一个现象:如果用广谱抗生素(ABX,万古霉素+新霉素+西司他丁)治疗肝癌模型小鼠来消灭某些细菌,会使小鼠的肝脏肿瘤减小减少,并且还降低了肿瘤从肝脏转移到他处的情况,这个现象依赖于NKT(自然杀伤细胞)的富集。已知NKT是通过CXCR6受体接收肝窦内皮细胞(LSECs)释放的CXCL16信号来杀死具有CD1d抗原分子表达的肿瘤细胞。研究人员验证发现,用抗生素处理过的小鼠肝脏中CXCL16含量确实显著增加。
根据文献和实验研究,肠道微生物对胆汁酸存在修饰作用(将初级胆汁酸转化为次级胆汁酸),而肝脏中CXCL16的表达水平与初级胆汁酸的水平呈正相关关系,与次级胆汁酸呈负相关。
到底是哪些肠道微生物起了作用,将初级胆汁酸代谢成为次级胆汁酸?研究人员用16S扩增子测序比较了抗生素使用前后的细菌差异表达谱,并结合靶向胆汁酸检测 (包括初级与次级胆汁酸) 结果,确定Clostridium species(梭菌属) 可以修饰初级胆汁酸变为次级胆汁酸。
最终探明的机制如下:Clostridium Species被杀死→阻断了胆汁酸的二级结构加工→阻断其运输到肝脏→促进了肝脏CXCL16表达→NKT细胞被招募→肝癌细胞被杀死或者转移被抑制。
参考文献:
1.在基于LC-MS的整体代谢组分析过程中,我们所遇到的问题、局限、优势和未来展望。 Gika H.G., et al. LC-MS-based holistic metabolic profiling. Problems, limitations, advantages, and future perspectives.J Chromatogr B Analyt Technol Biomed Life Sci. 2014 Sep 1;966:1-6. doi: 10.1016/j.jchromb.2014.01.054. Epub 2014 Feb 8.
2.代谢组学在人体肠道微生态研究中的机遇和挑战。Smirnov K.S., et al. Challenges of metabolomics in human gut microbiota research.Int J Med Microbiol. 2016 Aug;306(5):266-279. doi: 10.1016/j.ijmm.2016.03.006. Epub 2016 Mar 15.